چکیده
طی هزاره نخست پیش از دوران مشترک (BCE) ایل های عشایری مرتبط با فرهنگ ساکایی در استپ اوراسیا گسترش یافته ، و پهنه ای به وسعت بیش از 3500 کیلومتر را پوشاندند.
برای دریافتن روند های دموگرافیک پشت پرده گسترش فرهنگ ساکایی ما داده های ژنومی 8 نفر و یک مجموعه داده های متوکندریایی 96 نفر از شرق و غرب استپ اوراسیا را آنالیز کردیم.
استنتاج ژنومی آشکار می سازد که ساکاها در شرق و غرب استپ اوراسیا در بهترین شکل می توانند ترکیبی از اجزای نیایی مربوط به یامنایا و مؤلفه شرقی توصیف شوند.
مدل سازی دموگرافیک برای گروه های شرقی و غربی منشاء مستقل و جریان ژن مداوم بین آنها را نشان می دهد ، این یافته توضیحی برای شباهت های فرهنگ مادی چشمگیر این دو گروه است.
همچنین ما شواهدی دال بر اینکه جریان ژن قابل ملاحظه ای در اوایل عصر آهن از شرق به غرب اوراسیا صورت گرفته است یافتیم.
مقدمه
طی هزاره نخست پیش از میلاد جمعیت کوچ نشین در استپ اوراسیا از کوه های آلتای به فرای شمال دریای سیاه تا حوزه کارپات گسترش یافتند1.
بنا بر اولین اطلاعات در روایات هردوت (490 / 480-424 پیش از میلاد)2 ساکاهای کلاسیک که از سده هفتم پیش از میلاد در منطقه پونتیک شمالی زندگی می کرده اند ، شناخته شده ترین آن ها بوده اند.
تاریخ نگاران یونانی و فارسی در هزاره اول پیش از میلاد وقایع تاریخی ماساژت ها و سارومات ها را شرح داده اند ، و بعد ، سرمتی و ساکای: دارای آثاری مشابه آنچه در آثار تاریخی ساکای کلاسیک یافت شده است هستند ، آثاری مانند سلاح ، مطیع کردن اسب و آداب هنری بخصوص ‘سبک رسم حیوانات’.
بنابراین ، این گروه ها متعلق به فرهنگ ساکایی قلمداد می شوند و برای آن ها عنوان ‘ساکاها’ اطلاق می شود. ما نیز برای تسهیل در ادامه نوشته ، تمام گروه های عشایری عصر آهن استپ که معمولا با فرهنگ ساکایی در ارتباط هستند را ‘ساکا’ اطلاق خواهیم کرد.
منشاء فرهنگ گسترده ساکایی مدت طولانیست مورد مباحثه در باستان شناسی اوراسیاست. نخست تصور بر این بود که شمال دریای سیاه خاستگاه و مرکز فرهنگ ساکایی است3 ، تا اینکه ترنوزکین نظریه منشاء آسیای میانه را تدوین کرد4.
از سوی دیگر ، شواهدی مانند کورگان آرزهان1 در تووا5 که قدیمی ترین کورگان ساکاها5 محسوب می شود از منشاء اوراسیای شرقی حمایت می کند.
به وسیله تاریخ نگاری دفن در شرق و غرب اوراسیا مشخص شده است که کورگان های شرقی قدمت بیشتری نسبت به همتایان غربی خود دارند7،6. به اضافه ، عناصری مانند مشخصه ‘سبک رسم حیوانات’ متعلق به سده دهم قبل از میلاد4،1 یافت شده در منطقه رودخانه یئنی سئی و چین امروزی ، از حضور اولیه ساکاها در شرق حمایت می کند.
آثار و هنر ساکایی مدت کوتاهی پس از تکوین در منطقه وسیعی گسترش یافت ، اما درک از دینامیک واقعی جمعیتی که این فرهنگ را گسترش دادند ناچیز است.
مطالعات ژنتیکی جمعیت های آسیای میانه بر پایه DNA میتوکندری(mtDNA) 912،11،10، باستان8 و معاصر نشان می دهد که آسیای میانه از نظر تاریخی تقاطعی برای حرکت جمعیت ها از شرق به غرب و بلعکس بوده است.
ادعا شده جریان ژن از شرق به غرب اوراسیا از اوایل دوران پالئولیتیک13،14 و مزولیتیک15 شروع شده است ، و بلعکس از غرب به شرق در عصر برنز16.
یک پژوهش ژنومی اخیر17 بر نقش کوچ جمعیت مرتبط با فرهنگ یامنایا ها و آندرونوو ها به سمت شرق در طی عصر برنز تاکید کرده ، که پیرو آن آمیختگی قابل توجهی با آسیای شرقی رخ داده است.
بیشتر پژوهش های ژنتیکی درباره عشایر اواخر عصر آهن ، تحت نمونه کوچک محدود بودند ، آن هم محدود به یک گروه فرهنگی ، و یا فقط بر پایه آنالیز 22،21،20،19،18mtDNA.
در این پژوهش ، داده های از mtDNA نود و شش فرد مرتبط با فرهنگ ساکایی در مناطق جغرافیایی و بازه زمانی مختلف ، توالی و آنالیز شده است: بعلاوه ، داده های ژنومی 8 نفر از این افراد حصول و آنالیز شد (جدول تکمیلی 1).
از قسمت غربی استپ اوراسیا ، نمونه های یافت شده در قفقاز شمالی منسوب به دوره نخست ساکاها (سده هشتم تا ششم پیش از میلاد) ، ساکاهای کلاسیک از منطقه دن-ولگا (سده سوم پیش از میلاد) ، و سرمتی های اولیه از پکرووکا ، واقع در جنوب غربی اورال (سده پنجم تا دوم پیش از میلاد) را شامل می شود.
از قسمت شرقی استپ اوراسیا ، ما نمونه هایی از شرق قزاقستان منسوب به فاز زواکینو-چیلیکتا (سده نهم تا هفتم پیش از میلاد) را آنالیز کردیم؛ از سایت آرزهان2 منسوب به فرهنگ آلدی بل در تووا (سده هفتم تا ششم پیش از میلاد)؛ و از فرهنگ تاگار ناحیه مینوسینسک (سده پنجم پیش از میلاد).
اکثر نمونه های به وجود آمده در این پژوهش یا بازیابی شده از منابع علمی مربوط به سده های چهارم و سوم پیش از میلاد ، یافت شده از قسمت های مغولستان ، قزاقستان و روسیه کوهستان آلتای است.
یافته هایی که از این مناطق به دست آمدند همه متعلق به فرهنگ پازیریک بودند ، این فرهنگ پس از نخستین اکتشافات توسط گریازنوف در سال های 1927 و 1929 در دره پازیریک با این نام نامگذاری شد ، و با گور های غنی و محافظت شده با یخ خود شهرت یافته ، شرایطی که بدن های انسان ، آثار نقش دار و لباس به جا مانده به خوبی حفظ شده است25،24،23) شکل 1 .(
|
شکل 1: بازسازی یک ساکایی
یافت شده در كورگان اولون-كورین-گل 10 ، كوه های آلتای ، مغولستان (بازسازی توسط: دیمیتری پوزدنیاکف).
|
جمعیت ساکای شرقی و غربی که در اینجا مورد بررسی قرار می گیرند با فاصله ای 2000 تا 3500 کیلومتری از هم جدا شده اند ، ولی آثار باستانی نشانگر سبک زندگی و فرهنگ مشابه در آنها است.
اهداف این پژوهش بررسی میزان ارتباط ژنتیکی این گروه ها با یکدیگر خواهد بود و یا اینکه آیا منشاء مشترکی دارند ، و همچنین روشن سازی تاریخ دموگرافیک و رابطه ژنتیکی آن ها با جمعیت های معاصر.
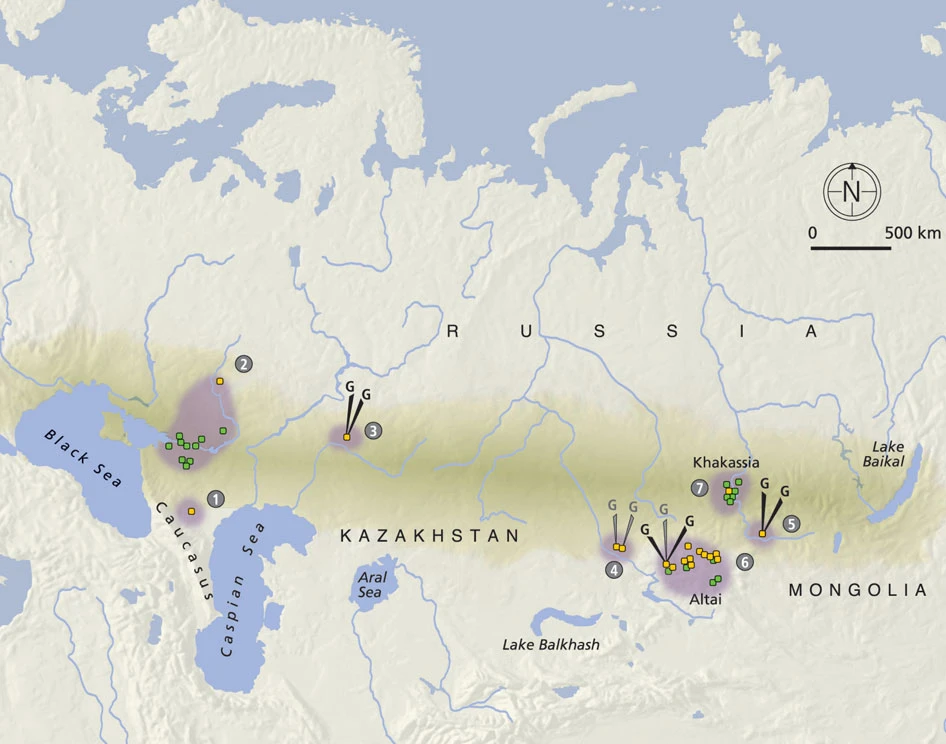
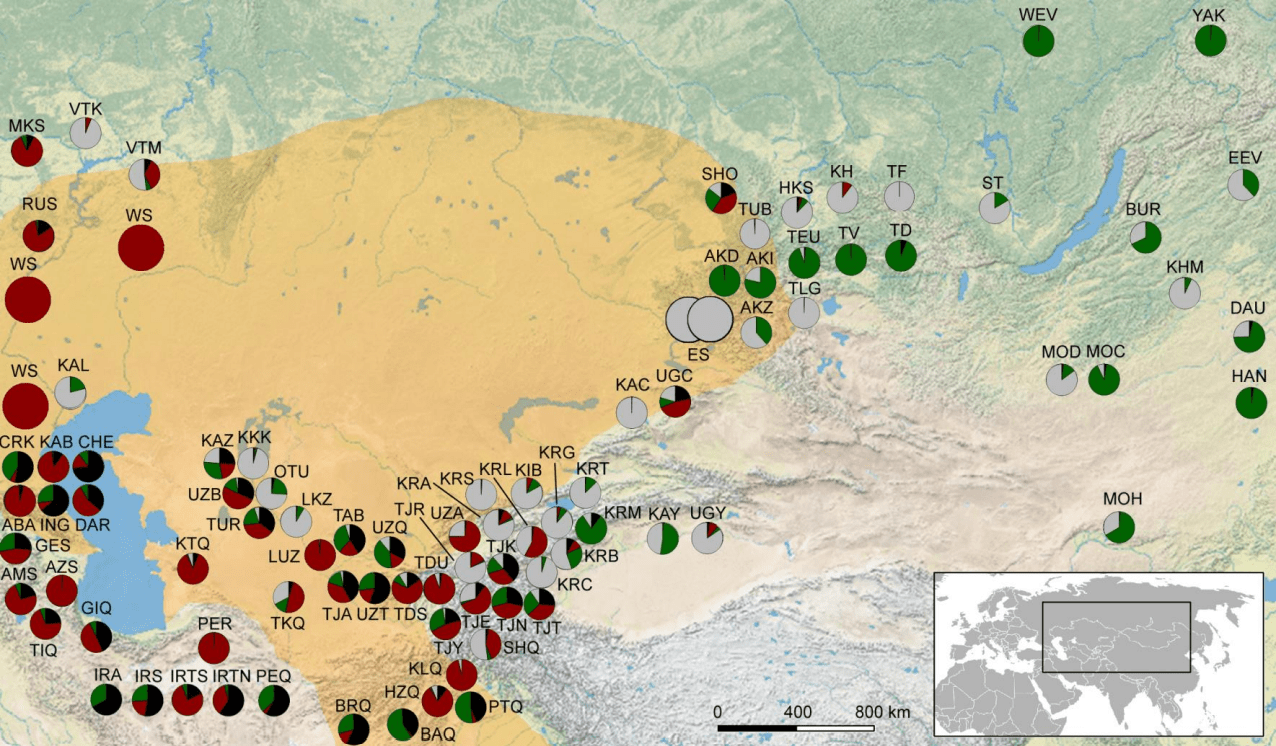
ما داده های عشایر عصر آهن به وجود آمده در این پژوهش و از منابع علمی (n=147 جدول تکمیلی 1) را بر اساس معیار های جغرافیایی و بازه زمانی در هفت گروه تقسیم بندی (شکل 2 را مشاهده کنید) و داده های جمعیتی گروه های افراد باستان را به همراه طیف گسترده ای از داده های 86 جمعیت معاصر (n=3,410 شکل تکمیلی 11 و جدول تکمیلی 19) از تمام اوراسیا را آنالیز کردیم.
آنالیز های ما شامل آرایه ای از روش های آماری و سلسله ای از راهبرد های استنتاج ژنتیکی جمعیت ، شامل مدل سازی صریح دموگرافیک است.
|
شکل 2: پراکندگی سایت های نمونه مورد آنالیز برای این پژوهش (زرد) شامل داده ها از منابع علمی (سبز).
اعداد گروه هاي تعريف شده را نشان مي دهند (#): غرب: 1# دوره اولیه ساكایی قرن هشتم تا ششم پیش از ميلاد (n=4) #2 مرحله ساکای کلاسیک قرن ششم تا دوم پیش از میلاد (n=19) #3 سرمتی اولیه قرن پنجم تا دوم پیش از میلاد (n=11) شرق: 4# فاز زواکینو-چیلیکتا قرن نهم تا هفتم پیش از میلاد (n=11) #5 فرهنگ آلدی بل قرن هفتم تا ششم پیش از میلاد (n=15) #6 فرهنگ پازیریک قرن چهارم تا سوم پیش از میلاد (n=71) #7 فرهنگ تاگار/تس قرن هشتم پیش از میلاد – قرن اول میلادی (n=16) فلش ها با G نمونه هایی را نشان می دهد که داده های ژنومی آنها حصول شده ، سیاه برای ظبط داده و خاکستری برای داده های تفنگ ساچمه ای ، منبع نقشه: cartomedia-Karlsruhe.
|
نتایج
نمونه ها و داده های توالی
ما داده های ژنومی را در یک مجموعه هدف از 1،233،553 27،26SNP برای 6 نفر ایجاد کردیم ، دو سرمتی اولیه از منطقه جنوبی اورال (PR9 ، PR3 گروه 3# در شکل 2 قرن پنجم تا دوم پیش از میلاد) ، دو فرد از برل در شرق قزاقستان (Be9 ، Be11, #6) متعلق به دوره پازیریک (قرن چهارم تا سوم پیش از میلاد) ، و دو فرد از کورگان آرزهان2 (A10, A17, #5) که به فرهنگ آلدی بل (قرن هفتم تا ششم پیش از میلاد) تعلق داشتند.
ما توسط تفنگ ساچمه ای NGS برای Be9 و دو فرد دیگر از شرق قزاقستان (Is2 و Ze6 گروه 4#) متعلق به فاز زواکینو-چیلیکتا (قرن نهم تا هفتم قبل از میلاد) ، پوشش کم (<0.3x) مجموعه های ژنوم کل را ایجاد کردیم (جدول 1 ، جداول تکمیلی 20 و 21).
جدول 1 لیست کلیه افراد باستان در این پژوهش که داده های هسته از آن ها حصول شده است.
| نمونه | مکان | فرهنگ | تاریخ | تعداد SNP که با آرایه منشاء انسانی همپوشانی دارند | پوشش تفنگ ساچمه ای | گروه نمونه ها از (شکل 2) |
| PR3 | پکرووکا ، روسیه | سرمتی اولیه | سده 5-2 ، پیش از میلاد | 306,498 | (غرب) 3 | |
| PR9 | پکرووکا ، روسیه | سرمتی اولیه | سده 5-2 ، پیش از میلاد | 186,890 | (غرب) 3 | |
| A10 | آرزهان ، روسیه | آلدی بل | سده 7-6 ، پیش از میلاد | 427,557 | (شرق) 5 | |
| A17 | آرزهان ، روسیه | آلدی بل | سده 7-6 ، پیش از میلاد | 108,952 | (شرق) 5 | |
| Be9 | برل ، قزاقستان | پازیریک | سده 4-3 ، پیش از میلاد | 549,958 | 0.30 | (شرق) 6 |
| Be11 | برل ، قزاقستان | پازیریک | سده 4-3 ، پیش از میلاد | 420,749 | (شرق) 6 | |
| Is2 | ایزمایلوو ، روسیه | زواکینو-چیلیکتا | سده 9-7 ، پیش از میلاد | 74,469 | 0.12 | (شرق) 4 |
| Ze6 | زواکینو ، روسیه | زواکینو-چیلیکتا | سده 9-7 ، پیش از میلاد | 163,338 | 0.28 | (شرق) 4 |
ما علاوه بر این توالی های صریح و قابل تکرار mtDNA را از ناحیه بسیار متغیر 1 (HVR1; bp 16,013–16,410) برای 96 نمونه (از 110 نمونه آنالیز شده) را به دست آورده (جدول تکمیلی 5) و برای 90/96 نمونه کد اضافی منطقه SNP تایپ کردیم (جدول تکمیلی 6).
رابطه ژنتیکی و منشاء گروه های ساکایی
گروه نمونه شرقی (n=113) را می توان در چهار زیر گروه فرهنگی تقسیم کرد که به ترتیب وقوع زمانی در هزاره اول قبل از میلاد پراکنده شدند (شکل 2). با آنالیز mtDNA ، هیچ فاصله ژنتیکی قابل توجهی بین این گروه ها پیدا نکردیم (جدول تکمیلی 8).
ما سپس برای تست پیوستگی بین گروه های ساکایی دوره های پیشین (4#: زواکینو-چیلیکتا و 5#: آلدی بل؛ n=26) و پسین (6#: پازیریک؛ n=71) در شرق از محاسبات تقریبی بیزی (ABC)28 استفاده کردیم.
گروه تاگار/تس (7#) به دلیل داده های غیر دقیق کنار گذاشته شد.
آنالیز ها نشان داد که آن ها مشتق شده از یک جمعیت واحد هستند ، و در بازه زمانی در نظر گرفته شده در اینجا در حال گسترش بودند ، به این معنا که ، این دو نمونه ممکن نیست نماینده دو جمعیت مستقل باشند که زودتر از 108 نسل پیش از حال (g BP) یا ∼2.7 ky BP واگرایی داشته اند.
این سناریو در روش انتخاب ما نیز بسیار پشتیبانی شد (جدول تکمیلی 9 رگرسیون لجستیک ، P=0.995 ، شبکه های عصبی ، P=0.568 ، مقایسه. اطمینان از مدل انتخاب شده جدول تکمیلی 10 ، پارامتر های مدل در جدول تکمیلی 11).
و دو سناریویی که دو نمونه گروه ساکای شرقی را مشتق شده از دو جمعیت که قبلا واگرایی داشته اند در نظر می گرفتند از پشتیبانی آماری بسیار کمی برخوردار هستند (تجمیع احتمالات پسین: رگرسیون لجستیک ، P<0.001 ، شبکه های عصبی ، P=0.001).
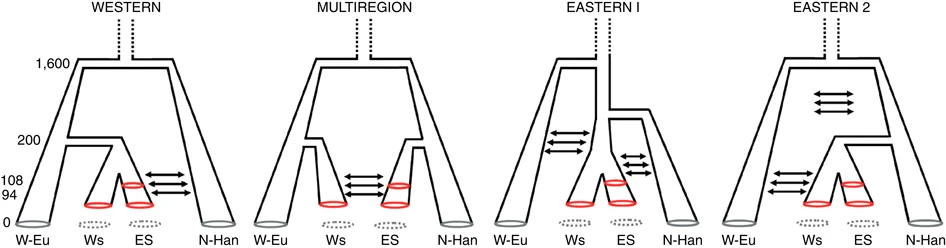
از آنجا که فاصله ژنتیکی بین گروه های ترکیبی ساکای شرقی در مقابل ساکای غربی نسبتا اندک است (FST=0.01733; P-value=0.02148±0.0045) ، ما برای بررسی بیشتر امکان داشتن منشاء مشترک ساکای شرقی و غربی از ABC استفاده کردیم (شکل 3).
برای این آنالیز نمونه هایی معاصر از نمایندگان تنوع ژنتیکی اوراسیا مشمول بودند (یادداشت تکمیلی 1). طبق الگوریتم انتخاب مدل ما ، یک مدل چند منطقه ای بهترین تناسب را با الگو های گوناگون تجربی مشاهده شده داشت. (جدول تکمیلی 12 ، نزدیک ترین شبیه سازی 0.5% ، احتمال پسین P=0.708 برای رگرسیون لجستیک ، P=0.715 برای روش شبکه های عصبی) ، در حالی که یک مدل از منشاء غربی نیز برخی پشتیبانی هایی داشت (جدول تکمیلی 12 ، نزدیک ترین احتمال 0.5% ، احتمال پسین P=0.286 برای رگرسیون لجستیک ، P=0.267 برای روش شبکه های عصبی).
بنابراین ، بر اساس آنالیز های ما نمی توان مدل منشاء غربی را در مقابل مدل منشاء شرقی کاملا نادیده گرفت. علاوه بر این ، مقایسه جفتی از طریق محاسبه فاکتورهای بیز ، امر قابل توجه قوی (رگرسیون لجستیک) یا ضعیف قابل توجه (شبکه عصبی) حمایت از منشاء چند منطقه ای نسبت به مدل منشاء غربی را نشان می دهد (مراجعه کنید به یادداشت تکمیلی 1 ، جدول تکمیلی 14 و شکل تکمیلی 5b).
این نشان می دهد شاید گروه های ساکای شرقی و غربی به صورت مستقل -شاید در ناحیه های مربوط به خود- به وجود آمده اند و پس از آن گسترش های قابل توجهی (در هزاره اول قبل از میلاد) در جمعیت تجربه کرده اند. مهم این که ، شبیه سازی های ما از جریان مداوم ژن بین گروه های ساکایی عصر آهن پشتیبانی می کند ، با نشانه هایی از جریان ژن نامتقارن به جای عکس آن از گروه های غربی به شرقی (برای اطلاعات بیشتر به جداول تکمیلی 13 و 15 مراجعه کنید).
| شکل 3: سناریو های احتمالی برای منشاء جمعیت ساکاها
اوراسیایی غربی: W-Eu ، گروه های ساکای غربی: WS ، گروه های ساکای شرقی: ES ، هان چین: N-Han اعداد سمت چپ به نسل های پیش از زمان حال منسوب هستند.
|
برای این که از نظر باستان شناسی حرکت های جمعیتی در سیتره آسیای میانه در عصر برنز غالبا با گسترش فرهنگ آندرونوو مرتبط است29 ، ما از ABC استفاده کردیم تا نمونه گروه های عشایری عصر برنز میانه از غرب سیبری که اکثر آن ها با فرهنگ آندرونوو مرتبط هستند را با مدل دموگرافیک ترجیحی برای منشاء ساکاها متناسب کنیم.
به این منظور -و بر اساس میزان کم FST بین این گروه ها- ما 40 نمونه مربوط به فرهنگ آندرونوو در استپ جنگلی سیبری غربی30 و 9 نمونه از همان فرهنگ در منطقه کراسنویارسک31 را که همه آن ها متعلق به نیمه اول هزاره دوم پیش از میلاد بودند را ترکیب کردیم.
نتایج بررسی از پیوند بسیار قوی بین گروه های عصر برنز میانه و ساکاهای شرقی حمایت می کند (جداول تکمیلی 16 و 17).
با این حال ، این شبیه سازی ها نتوانستند الگوهای تنوع ژنتیکی ملاحظه شده در جمعیت عصر برنز را به طور کامل ظبط کنند ، این نشان می دهد که تاریخ دموگرافیک واقعی تبار جمعیت عصر آهن ممکن است پیچیده تر از آن چیزی باشد که در این مقاله در نظر گرفته شده است (برای اطلاعات بیشتر به یادداشت تکمیلی 1 و 32 مراجعه کنید).
تنوع ژنتیکی و تبار گروه های ساکایی
هاپلوگروپ هایی که در عشایر عصر آهن یافت می شوند در جمعیت های معاصر در شرق (A, C, D, F, G, M, Y, Z) و غرب (HV, N1, J, T, U, K, W, I, X) اوراسیا غالب هستند. تنوع هاپلوتیپ میتوکندری در نمونه های ما از محدوده 0.958±0.036 در تاگار/تس (گروه 7# در شکل 2) تا 1.000±0.039 در سرمتی اولیه (3#؛ جدول تکمیلی 7) است.
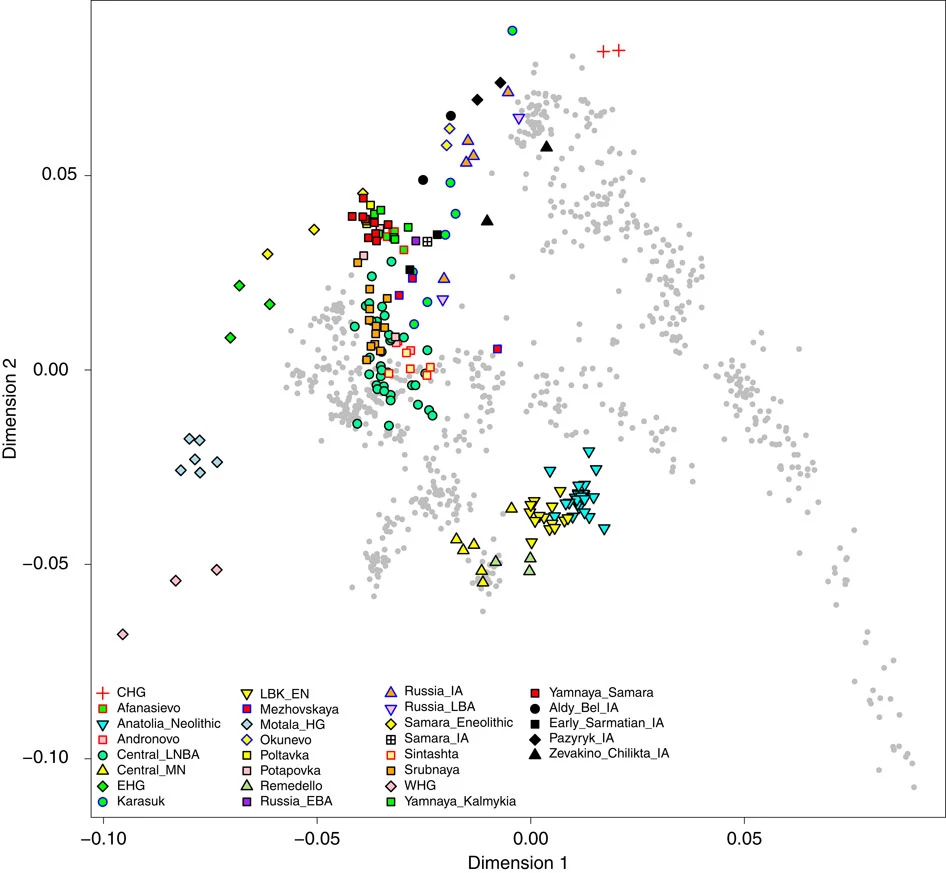
با استفاده از داده های SNP هسته ، ما آنالیز مؤلفه اصلی33 (PCA) را با 777 اوراسیایی غربی معاصر35،34،26 و هشت نمونه ساکای عصر آهن تازه گزارش شده و همچنین 167 نمونه باستانی دیگر از اروپا ، قفقاز و سیبری از منابع علمی36،34،17 به انجام رساندیم (شکل 4).
دو نمونه سرمتی اولیه از غرب (گروه 3# در شکل 2) نزدیک به یک نمونه عصر آهن از ناحیه سامارا34 قرار دارند و بطور کلی به نمونه های یامنایا مربوط به عصر برنز اولیه از سامارا34 ، قالمیقستان17 و نمونه های پولتاوکا عصر برنز میانه از سامارا34 نزدیک هستند.
نمونه های شرقی از پازیریک (6#) ، آلدی بل (5#) و زواکینو-چیلیکتا (4#) بخشی از یک خوشه سست همراه با نمونه های دیگر از آسیای میانه17 ، از جمله آن هایی که از اوکونوو ، نمونه های اواخر عصر برنز ، نمونه های عصر آهن روسیه و کاراسوک هستند.
این نمونه ها در تضاد با نمونه های قبلی استپ اوراسیا متعلق به گروه های آندرونوو17 ، سینتاشتا17 و سروبنایا34 که با افراد اواخر عصر نوسنگی/عصر برنز از قاره اروپا35،34 همپوشانی دارند و در طرح PCA در پایین سمت کشاورزان اولیه اروپا و آناتولی34 قرار میگیرند هستند.
|
شکل 4: آنالیز مؤلفه اصلی
PCA افراد باستان (مطابق رنگ ها اختصار ها و شرح را ببینید) بر داده های افراد معاصر اوراسیای غربی (خاکستری). افراد ساکایی به رنگ سیاه هستند. شکارچی گرد آورنده قفقاز: CHG ، اواخر نو سنگی/عصر برنز: LNBA ، نو سنگی میانه: MN ، شکارچی گرد آورنده اروپای شرقی: EHG ، لینربندکرامیک اوایل عصر نو سنگی: LBK_EN ، شکارچی گرد آورنده: HG ، اوایل عصر برنز: EBA ، عصر آهن: IA ، اواخر عصر برنز: LBA ، شکارچی گرد آورنده غربی: WHG.
|
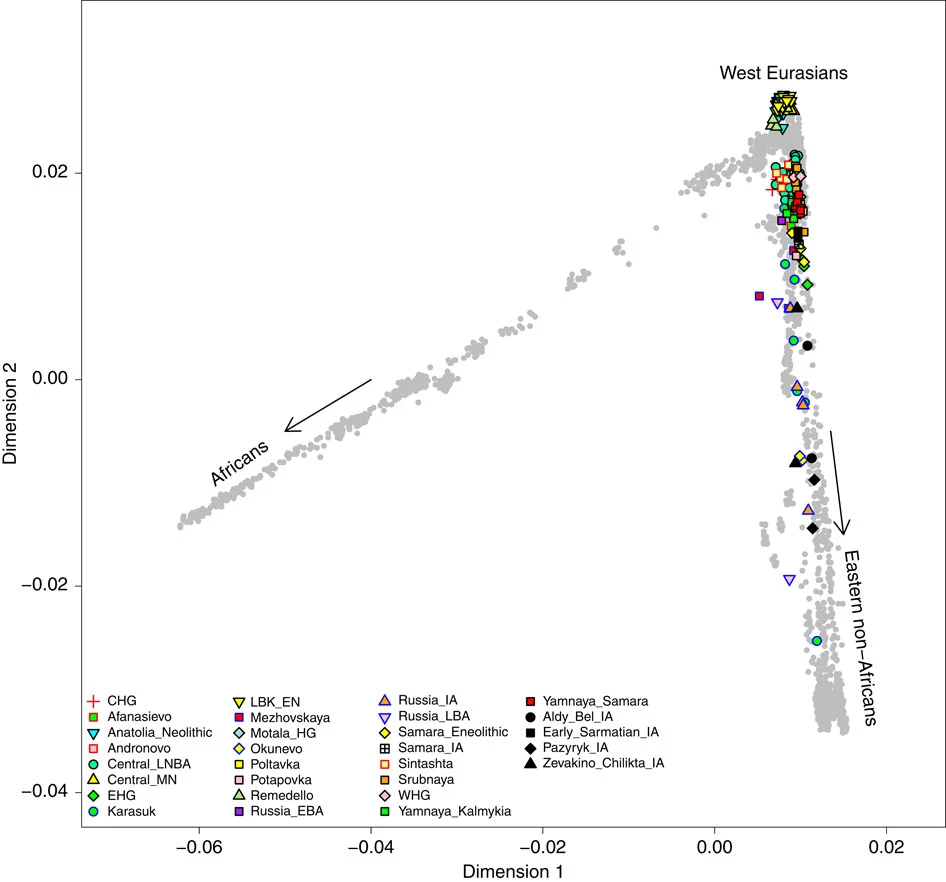
از آنجا که PCA اوراسیای غربی در شکل 4 امکان بررسی نمونه های باستانی در رابطه با جمعیت های معاصر اوراسیای شرقی را نمی دهد ، ما PCA تمام 2345 فرد معاصر در مجموعه داده های منشاء انسانی35 را به عمل آوردیم ، و افراد باستانی را نیز در آن گنجاندیم (شکل 5).
از این PCA آشکار است كه تبار نمونه های عصر آهن در زنجیره بین اوراسیاییان غربی و شرقی غیر آفریقایی معاصر جای می گیرد ، كه با آنالیز های هاپلوگروپ میتوکندری همخوانی دارد.
ساکاهای شرقی تقریبا میزان تبار mtDNA مشترک برابر از شرق و غرب اوراسیا را نشان می دهند ، در حالی که گروه های ساکای غربی فرکانس تبار کم تری نسبت به ساکای شرقی را نشان می دهند ، و حتی این میزان در چهار نمونه از مرحله اولیه ساکاها قرن هشتم تا ششم پیش از میلاد (گروه 1# در شکل 2) به صفر رسیده ، و در دوره های بعدی (قرن ششم تا دوم پیش از میلاد؛ 2# و 3#) به 18-26٪ می رسد (جدول تکمیلی 7).
|
شکل 5: آنالیز مؤلفه اصلی
PCA افراد باستان (مطابق رنگ ها اختصار ها و شرح را ببینید) بر داده های افراد معاصر مجموعه منشاء انسانی (خاکستری). افراد ساکایی به رنگ سیاه هستند. شکارچی گرد آورنده قفقاز: CHG ، اواخر نو سنگی/عصر برنز: LNBA ، نو سنگی میانه: MN ، شکارچی گرد آورنده اروپای شرقی: EHG ، لینربندکرامیک اوایل عصر نو سنگی: LBK_EN ، شکارچی گرد آورنده: HG ، اوایل عصر برنز: EBA ، عصر آهن: IA ، اواخر عصر برنز: LBA ، شکارچی گرد آورنده غربی: WHG.
|
آمار-f
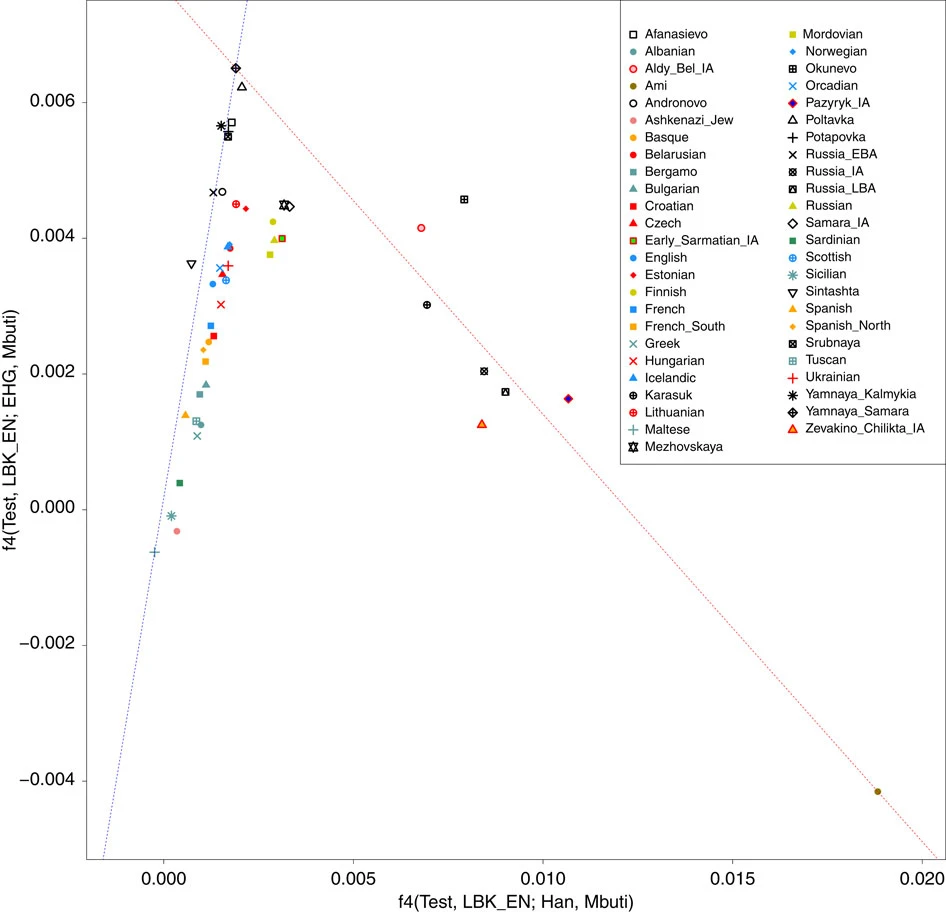
ما از آمار های f4 فرم f4(Test, LBK; EHG, Mbuti) و f4(Test, LBK; Han, Mbuti) استفاده کردیم ، که به ترتیب صفر برای آن نمونه هایی که کلاد با LBK ، و مثبت برای جمعیت هایی که نیای مربوط با EHG- یا هان را دارا هستند است.
ما نتایج را در برابر یکدیگر نمودار کردیم ، که منجر به یک الگوی V شکل شد ، و یامنایا در راس قرار گرفت (شکل 6).
ساکاهای عصر آهن در امتداد خطی از یامنایا تا امی (یک جمعیت از تبار آسیای شرقی که هیچ ترکیب جمعیتی را تجربه نکرده است) قرار می گیرند ، که سازگار با داشتن تبار از جمعیتی مشابه از نظر ژنتیکی با این دو گروه است.
|
شکل 6: تجسم نتیجه آمار-f
مقادیر f4(Test, LBK; Han, Mbuti) در محور x و مقادیر f4(Test, LBK; EHG, Mbuti) در محور y ترسیم می شوند ، انحراف مثبت از صفر انحراف از یک کلاد بین Test و LBK را نشان می دهد. یک خط چین قرمز بین یامنایا از سامارا و امی ترسیم شده است. جمعیت های عصر آهن که می توانند به عنوان ترکیبی از یامنایا و اوراسیای شرقی (مانند امی) مدل شود در اطرف این خط قرار گرفته اند ، و به نظر می رسد با کلاین اصلی شمال/جنوب اروپا (آبی) در سمت چپ بر روی محور x متفاوت است.
|
ما همچنین مقادیر فرم f3(Test; Yamnaya_Samara, Han) را نیز محاسبه کردیم ، تا بررسی کنیم آیا جمعیت Test فرکانس های آلل میانی بین Yamnaya_Samara و Han را داراست ، این روش به عنوان پراکسی برای جمعیت های منشاء ممکن استفاده می شود.
فرکانس های آلل میانی تنها زمانی اتفاق می افتند که جمعیت test ترکیبی از جمعیت هایی مربوط به این دو منشاء37 باشد. این آمار برای تمامی ساکاها قابل توجه منفی است و این نشان می دهد آمیختگی حادث شده است (شکل تکمیلی 13).
آنالیز های آمیختگی
ما آنالیز آمیختگی39،38 را از 2345 انسان معاصر35 ژنوتیپ بر آرایه منشاء انسانی37،35 و 175 فرد باستانی در مجموعه ای از 296،340 SNP که در تلاقی با آنهایی که در مجموعه منشاء انسانی قرار دارند انجام دادیم.
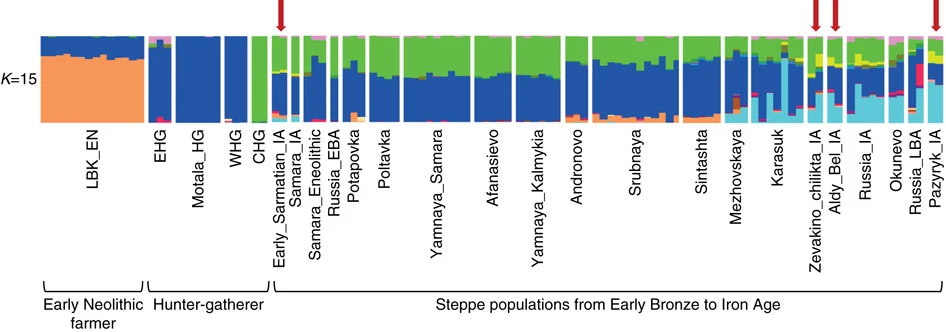
نتایج برای افراد باستان در شکل 7 برای K=15 نشان داده شده ، که احتمالا دارای بیش ترین مقدار لگاریتم درست نمایی است (آنالیز های کامل در شکل تکمیلی 14 قابل دسترسی اند).
همه جمعیت های استپی دارای اجزایی هستند که در شکارچی گرد آورنده اروپایی (آبی) ، و در شکارچی گرد آورنده قفقاز از گرجستان36 (سبز) در حداکثر است.
همچنین یک زیر مجموعه از جمعیت استپ (از جمله سروبنایا ، سینتاشتا و آندرونوو) دارای تبار از کشاورزان اولیه (نارنجی) هستند ، حال آنکه زیر مجموعه ای متفاوت (شامل تمام نمونه های عصر آهن) از اجزای (آبی روشن) برخوردار هستند که در نگاناسان (مردم سموئید از شمال سیبری) در حداکثر است ، و در بین جمعیت های گوناگون معاصر از سیبری و آسیای میانه گسترده است.
بعلاوه ، نمونه های عصر آهن عنصر تباری را نشان می دهند که در جمعیت های آسیای شرقی (زرد) در حداکثر است ، نوعی تبار در سطح پایین -محدود- که بین ساکنان اولیه استپ وجود دارد ، که با مشاهدات از PCA و آمار-f درباره اینگونه از آمیختگی ها همخوانی دارد.
|
شکل 7: نتایج آمیختگی برای مردمان باستان
فلش های قرمز افراد ساکایی عصر آهن مورد بررسی را نشان می دهند. لینربندکرامیک اوایل عصر نو سنگی: LBK_EN ، شکارچی گرد آورنده اروپای شرقی: EHG ، شکارچی گرد آورنده از موتالا (سوئد): Motala_HG ، شکارچی گرد آورنده غربی: WHG ، شکارچی گرد آورنده قفقاز: CHG ، عصر آهن: IA ، اوایل عصر برنز: EBA ، اواخر عصر برنز: LBA.
|
مدل سازی جمعیت های استپ باستانی
ما جمعیت های استپ را به عنوان ترکیبی از یامنایا اوایل عصر برنز و کشاورزان LBK از اروپای مرکزی یا آسیای شرقی (که توسط چینی های هان نمایندگی می شود) مدل کردیم.
ما روش qpWave/qpAdm استفاده شده در هاک و همکاران26 که یک آمار تست برای تعداد جریان تبار به یک جمعیت Test ارائه می دهد و تخمین نسبت آمیختگی را ممکن می سازد را اعمال کردیم.
ما در اپلیکیشن از 5 برون گروه: Onge ، Papuan ، 42MA1 ، 41Kostenki14 ، 40Ust_Ishim استفاده کردیم.
ابتدا ما محاسبه کردیم که آیا Test و یامنایا از سامارا می توانند از جریان تبار مشترکی باشند.
در قدم بعد تست های ما شامل کشاورزان LBK است که آیا Test ، یامنایا از سامارا و کشاورزان LBK از اروپای مرکزی می توانند از دو جریان تباری باشند ، اگر چنین باشد Test می تواند به عنوان ترکیبی از دو جمعیت دیگر مدل شود.
نتایج ما نشان می دهد که ساکاهای عصر آهن و یامنایا از یک جریان تبار واحد نیستند (جدول تکمیلی 23) ، همچنین نمی توانند به عنوان ترکیبی از یامنایا و LBK مدل شوند (جدول تکمیلی 24).
به این خاطر ما یک الگوی دیگر که در آن یامنایا و هان اجزای ترکیب بودند در نظر گرفتیم (جدول تکمیلی 25). این مدل با تمام گروه های ساکایی تناسب داشت ، متناسب با گروه هایی که تبار مربوط به شرق که در جمعیت های دیگر یافت نمی شوند دارند.
همچنین گروه های ساکایی عصر آهن می توانند به عنوان ترکیبی از مؤلفه های نگاناسان شمال سیبری و یامنایا مدل شوند (یادداشت تکمیلی 2 ، جدول تکمیلی 26).
نوادگان ساکاهای عصر آهن
طرح مقیاس گذاری چند بعدی (MDS) بر اساس مسافت های رینولدز (شکل تکمیلی 1) نشان داد جمعیت ساکای شرقی و غربی در استپ اوراسیا از نظر ژنتیکی به یک دیگر نزدیک تر هستند تا جمعیت معاصر این منطقه.
همچنین آنالیز های AMOVA صورت گرفته برای گروه های معاصر و باستانی استپ شرقی و غربی حمایت بیشتری از این یافته داشت.
ما نسبت FCT بیشتری را بین جمعیت های معاصر شرق و غرب (FCT=0.0835) نسبت به جمعیت باستانی (FCT=0.0262) مناطق مشابه یافتیم.
یک آزمایش استمرار بین دو گروه عصر آهن (‘غرب’ و ‘شرق’) و مجموعه بزرگی از جمعیت معاصر اوراسیا (n=86 ، جدول تکمیلی 19) به عمل آمد.
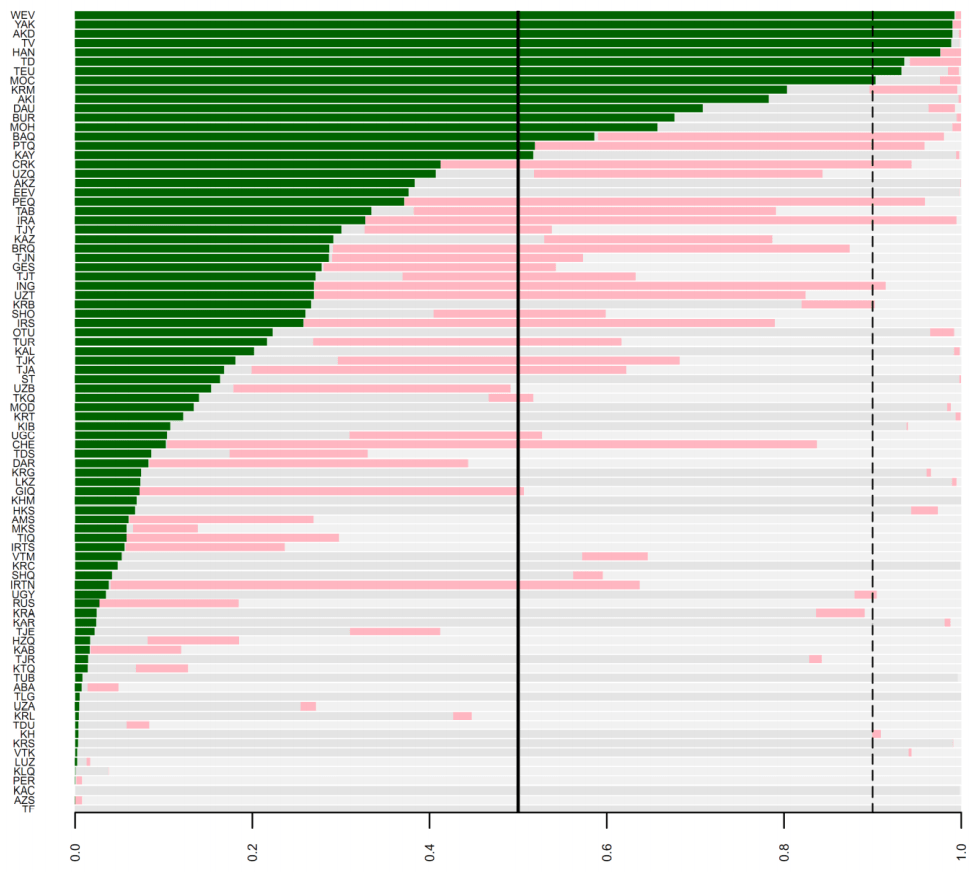
جمعیت های معاصری که دارای حمایت بالای آماری برای ارتباط ژنتیکی با ساکای غربی بودند در جغرافیایی غالبا نزدیک حضور دارند ، در حالیکه جمعیت های معاصری که دارای حمایت بالای آماری برای تبار از ساکای شرقی بودند در جغرافیای گسترده تر توزیع شده اند.
جمعیت های معاصر مرتبط با مردمان استپ غربی عصر آهن را می توان در گروه های قومی مختلف قفقاز ، روسیه و آسیای میانه (بین بسیاری از ایرانی ها و گروه های هندو-اروپایی زبان دیگر) یافت ، در حالیکه جمعیت هایی که دارای تشابه ژنتیکی با ساکای شرقی اند تقریبا به طور انحصاری بین گویندگان زبان ترکی یافت می شوند (شکل های تکمیلی 10 و 11).
مارکر های فنوتیپی
آلل های مشتق شده پیگمنتیشن مارکر تحت انتخاب در اروپایی ها در ساکای شرقی و غربی موجود اند ، از جمله افرادی که برای آلل های مشتق شده در SNP های انتخاب شده در SLC24A5 ، HERC2 یا SLC45A2 هموزیگوس هستند (منابع: 43 ، 44 ، 45).
در دو لوکوس LCT مرتبط با پایداری لاکتاز ، آلل های مشتق شده تنها در هتروزیگوت ها دیده می شود ، فقط در نمونه های ساکای شرقی ، و در فرکانس پایین (2–3%).
آلل های نیایی در ADH1B rs3811801 و rs1229984 تقریباً در مجموعه داده های ساکاها ثابت هستند ، مانند آنجه در اروپایی های معاصر است (آلل های مشتق شده ای که دارای مقاومت در برابر الکلیسم اند از آسیای شرقی تحت انتخاب هستند47،46).
ما آلل مشتق شده را در rs3827760 در ژن EDAR یک فرد پازیریک (گروه 6# در شکل 2) مشاهده کردیم. این آلل مشتق شده EDAR که مربوط به مورفولوژی دندان و مو است ، در آسیای شرقی معاصر دارای فرکانس بالا (87%)48 ، و در اروپایی های معاصر بسیار نادر است (∼1%)48. هر چند که در شکارچی-گردآورنده ماقبل تاریخ از سوئد (7.9–7.5 kya)34 نیز مشاهده شده است.
پس ، نتایج بررسی SNP های فنوتیپی نشانگر تفاوت های فرکانس بین اروپا و شرق آسیاست که سازگار با جریان ژن در عرصه استپ است.
مباحثه
نتایج بررسی های ما نشان داد گروه های عصر آهن -که مدت ها اعتقاد بر این بود از طریق آثار باستانی و فرهنگی با ساکاهای کلاسیک منطقه پونتیک شمالی مرتبط هستند- ارتباط ژنتیکی نیز دارند.
آنالیز های ABC ما استمرار جمعیت در ساکای شرقی پیش از هزاره اول قبل از میلاد و مقادیر کم FST بین گروه های ساکایی شرقی و غربی را آشکار می سازد.
گرچه آنالیز های ABC سناریو های مختلفی را برای منشاء ساکاها می سنجد ، در این حال سناریوی منشاء چند منطقه ای بیشترین حمایت را دارد ، با گروه های شرقی و غربی که به صورت مستقل در ناحیه مربوط به خود موجودیت یافتند.
با وجود منشاء مجزا و جدایی عظیم جغرافیایی ، مدل سازی دموگرافیک بر جریان ژن مداوم و قابل توجه بین گروه های شرقی و غربی اشاره می کند. که یک مکانیزم دموگرافیک محتمل و توضیحی برای مقادیر کم FST و یکنواختی کلی فرهنگ مادی ساکاها درست در گستره استب اوراسیا ارائه می دهد.
آنالیز های ژنومی ما آشکار می سازد که ساکنان غربی و شرقی استب دارای تبار از اوراسیای شرقی با درجات متفاوت هستند.
در آنالیز های آمیختگی ما ، یک مؤلفه تبار از آسیا شرقی در K=15 در تمام نمونه های عصر آهن پیدا میکنیم که در جمعیت های قبل عصر برنز در قسمت های غرب و شرق استپ اوراسیا نیافته بودیم.
یکی دیگر از مؤلفه های نیایی در جمعیت نگاناسان شمال سیبری به حداکثر رسیده و از هزاره دوم قبل از میلاد به بعد در استپ شرقی (اوکونوو ، کاراسوک ، مژوفسکایا) پدیدار می شود. سپس این مؤلفه نیایی در همه جمعیت های عصر آهن ظاهر می شود اما دارای سطح بالاتری در استپ شرقی نسبت به غربی است.
این یافته ها سازگار با پدیدار شدن تبار میتوکندری اوراسیای شرقی در ساکاهای غربی طی عصر آهن است ، و بر جریان ژن یا کوچ از طریق کمربند استپ اوراسیا حامل تبار شرقی/شمال سیبری از شرق به غرب تا منطقه دن-ولگا در جنوب روسیه دلالت دارد.
به طور کلی ، چنین به نظر می رسد که جریان ژن بین شرق و غرب اوراسیا در عصر آهن بیشتر و شدیدتر از دوران معاصر بوده است ، و این با جمعیت های عصر آهن استپ اوراسیا که گروه هایی نیمه عشایری اسب سوار بسیار متحرک بوده اند متناسب است.
در شرق ، ما یک آمیختگی متعادل از نسب های میتوکندریایی می یابیم که امروزه عمدتا در غرب اوراسیا یافت می شوند ، من جمله نسبت قابل توجه از نسب های شکارچی گرد آورنده ماقبل تاریخ ، و نسب هایی که در آسیای مرکزی و شرقی معاصر دارای فرکانس بالا اند ، که قبلا در اولین افراد عصر آهن مربوط به سده نهم تا هفتم پیش از میلاد و حتی پیش تر در یک نمونه mtDNA از عصر برنز مغولستان49 بودند.
همچنین نوعی نسب mtDNA اوراسیای غربی نیز در حوضه تاریم16 و قزاقستان8 وجود دارد که حتی در ناحیه کراسنویارسک در هزاره دوم پیش از میلاد31 نیز غالب بوده اند.
این الگو به فرایند آمیختگی بین جمعیت های شرقی و غربی اوراسیا اشاره می کند که در دوره های اولیه ، مطمئناً قبل از هزاره اول پیش از میلاد50،13 شروع شده است ، یافته ای سازگار با پژوهش اخیر نشان می دهد که حاملان فرهنگ یامنایا از لحاظ زنتیکی قابل تشخیص از جمعیت های فرهنگ آفاناسیو از منطقه آلتای-سایان نیستند.
این نشانگر این است که حاملان فرهنگ یامنایا نه تنها به اروپا26 بلکه به شرق نیز کوچ کرده و ژن های اوراسیای غربی -همچنین به طور بالقوه زبان های هندو-اروپایی- را به این منطقه17 منتقل کرده اند.
همه این مشاهدات مدرکی به میان می گذارد که الگوی ژنتیکی مرسوم به سادگی مدل انزوا-توسط-فاصله را دنبال نمی کند ، بلکه نشانگر جریان ژن قابل توجهی در مسافت های زیاد است.
همه افراد عصر آهن که در این پژوهش مورد بررسی قرار گرفته اند شواهد ژنومی برای داشتن نسب از شکارچی گرد آورنده قفقاز و شکارچی گرد آورنده اروپای شرقی را نشان می دهد. این با چنین عقیده ای که آمیختگی عناصر EHG و قفقازی در ناقلین فرهنگ یامنایا در استپ اروپا صورت گرفته و به آسیای میانه و سیبری26 منتقل شده همخوانی دارد. همه آنالیز های ما از فرضیه ای که ترکیب ژنتیکی ساکاها را به بهترین شکل ترکیبی از نسب مربوط به عناصر یامنایا و شرق آسیا/شمال سیبری به حساب می آورد حمایت می کند.
در خصوص بازماندگان عشایر عصر آهن ، ما پی بردیم جمعیت های معاصری که دارای رابطه نزدیک ژنتیکی با گروه های ساکایی اند در جغرافیایی نزدیک به سایت های دفن نمونه برداری شده هستند ، که نشانگر میزان استمرار جمعیت ها در دوره های تاریخی است.
نوادگان معاصر گروه های ساکای غربی در میان گروه های مختلف قفقاز و آسیای میانه یافت می شوند ، ولی با اینکه شباهت ها با ساکای شرقی بسیار گسترده تر است ، اما تقریبا به طور انحصاری بین گروه های ترک زبان (سابقا) عشایری ، بخصوص از شاخه قبچاق زبان ترکی یافت می شوند (یادداشت تکمیلی 1).
پیوند ژنتیکی بین ساکاهای شرقی و گویندگان زبان ترکی نیاز به بررسی بیشتر دارد ، بخصوص با این که تصور می شد که گسترش زبان های ترکی بسیار جدیدتر -یعنی ، از سده ششم میلادی به بعد- و بواسطه فرایند گسترش منتخبان به وقوع پیوسته است.
به طور بالقوه فاکتور های دموگرافیک بسیاری در منشاء تبار گویندگان زبان ترکی دخیل هستند ، مانند امواج مهاجرت مرتبط با شیونگ نو ، ترک های باستانی یا جمعیت های اولیه مغول.
میزان مشارکت ساکای شرقی در شکل گیری اولیه جمعیت ترک زبان می تواند توسط بررسی های ژنومی آینده مربوط به دوره های تاریخی پس از ساکاها مشخص شود.
روش ها
مواد نمونه ها
برای این پژوهش ، متریال نمونه اسکلت های انسان از مناطق مختلف قزاقستان و روسیه مبتنی بر ارتباط مجموعه باستان شناسی با آداب دفن و آثار ساکایی انتخاب شد.
ما 110 نمونه اسکلت را آنالیز کردیم.
که 5 نمونه بدون DNA ، و هشت نمونه دارای نتایج ضعیف بودند ، و فقط برای یک منطقه کدگذاری SNP ها قابل حصول بود.
در نهایت ، ما می توانستیم از 96 نمونه برای آنالیز های mtDNA استفاده کنیم.
هشت فرد را به کتابخانه ایلومینا تبدیل کردیم؛ و شش نفر برای ظبط ژنوم استفاده شدند ، همچنین دو مورد برای تعیین توالی تفنگ ساچمه ای ، و یک مورد نیز هم برای ظبط و هم برای تعیین توالی تفنگ ساچمه ای استفاده شد.
بطور کلی حفظ DNA به طور قابل ملاحظه ای خوب بود ، همراه با تغیرات جزئی که مربوط به منطقه یا مکان نمونه ها می شد (جداول تکمیلی 1 و 2).
آماده سازی نمونه ها
مراحل آماده سازی نمونه pre-PCR در یک پالودگاه که جدا از آزمایشگاه های post-PCR بود انجام شد.
آماده سازی نمونه ، استخراج DNA ، تقویت هر قطعه mtDNA و تقویت لوکوس هسته ، همان طور که قبلا شرح داده شده است انجام شد45.
برای آنالیز های mtDNA سی منطقه کد گذاری که سی و دو SNP هاپلوگروپ خاص ژنوم میتوکندریایی را پوشش می دهد از منابع علمی انتخاب شد53،52،51.
سیستم های مبادی اولیه (جدول تکمیلی 3) به وسیله PrimerSelect که بخشی از پکیج نرم افزاری (Lasergene (DNASTAR است طراحی شدند.
برای جلوگیری از تداخل قطعه ها تقویت در سه واکنش مختلف مولتی پلکس انجام شد (جدول تکمیلی 3).
PCR های مولتی پلکس با حجم کلی 40 میکرولیتر ، با استفاده از 20 میکرولیتر Qiagen Multiplex Kit Master Mix (Qiagen, Hilden, Germany) ، و 11 میکرولیتر ddH2O ، و 0.2 میکرولیتر از هر مبادی اولیه و 4 میکرولیتر عصاره DNA انجام شد.
وضعیت PCR مولتی پلکس به شرح زیر است:
نخستین دناتوراسیون به مدت 15 دقیقه در دمای 95 درجه سانتیگراد و به دنبال آن 36 چرخه از 40 دناتوراسیون در دمای 94 درجه سانتیگراد ، پختن به مدت 40-90 ثانیه در دمای 56 درجه سانتی گراد ، نگه داشتن به مدت 40-90 ثانیه در دمای 72 درجه سانتیگراد و برای ایجاد برخی واکنش ها نیز نگه داشتن نهایی به مدت 10 دقیقه در دمای 72 درجه سانتیگراد اضافه شد.
از PCR استاندارد برای تقویت قطعه هایی که در واکنش های مولتی پلکس موفق نبودند و برای تقویت HVR1 نیز از برخی نمونه های اضافی استفاده شد.
آنالیز های توالی و تصدیق
برای اجازه به تعیین توالی موازی 37 قطعه mtDNA تا 60 نمونه در دستگاه 454 GS FLX ، بارکد ها با پروتکل برچسب گذاری Meyer 2008 به قطعه های DNA مولتی پلکس شده پیوند داده شدند (منبع: 54) ، با این استثناء که مجموع حاصل PCR برای مرحله نخست مورد استفاده قرار گرفته است.
سه PCR مولتی پلکس از یک نمونه طی نخستین قدم پالایش ادغام شدند.
برای همه مراحل پالایش از کیت Qiagen MinElute PCR purification استفاده شد.
تهیه و تعیین توالی 454 کتابخانه توسط GATC Biotec AG در کنستانتس انجام شد.
حاصل های PCR منفرد بوسیله آنالیز کننده توالی ABI 3130 بعد از تعیین توالی چرخه Sanger استاندارد بوسیله کیت تعیین توالی چرخه BigDye Terminator v1.1 (Applied Biosystems, Life Technologies, Darmstadt, Germany) آنالیز شد.
برای مارکر های هسته توالی های زائد بوسیله CD-HIT-454 فرو ریخت (منبع: 55) ، خوشه های توالی منفرد حذف و توالی های خاص با 56mafft برای هر مارکر به توالی مرجع تراز و آلل ها شمارش شدند.
برای توالی های mtDNA توالی اجماع با SeqMan از پکیج DNASTAR Lasergene ساخته شد ، هاپلوگروپ ها با HaploGrep اختصاص داده شد ، که یک نرم افزار تحت وب است که از phylotree build 15 استفاده می کند (منابع: 57 ، 58) ، (جداول تکمیلی 4 و 6).
برای تصدیق نتایج ، باید هر موقعیت از HVR1 حداقل با سه توالی غیر مبهم از PCR های مستقل پوشانده می شد.
قطعات منطقه کدگذاری حداقل دو بار با 454 FLX توالی شدند.
تکثیر مستقل برای 22 نمونه توسط الکساندر پیلیپنکو در انیستیتو سیتولوژی و ژنتیک ، شعبه سیبری ، آکادمی علوم روسیه صورت گرفت.
تمام نتایج حاصله مطابق با نتایج به دست آمده در آزمایشگاه پالئوژنتیک ، در ماینتس بود.
طی مراحل آماده سازی نمونه ، کنترل های خالی در مراحل پودر سازی ، استخراج و تقویت قرار گرفتند.
میزان آلودگی کلی 2.2% بود.
آلودگی ها مشاهده و طی واکنش همان مراحل با توالی نمونه ها مورد مقایسه قرار گرفتند.
هیچ ارتباطی یافت نشد و از آنجا که توالی نمونه ها در واکنش های مستقل تکثیر می شوند همه نتایج تصدیق شدند.
آنالیز آمار زیستی
در این پژوهش برای آنالیز ژنتیکی جمعیت توالی های HVR1 از موقعیت 16,040–16,400 از 96 نمونه آنالیز شده برای این پژوهش و 51 نمونه اضافی از منابع علمی (جدول تکمیلی 1) استفاده شد.
از نرم افزار 59Arlequin 3.5.1.3 برای AMOVA و محاسبه تنوع ژنی (تنوع هاپلوتیپ)60 ، تنوع نوکلئوتید61،60 ، مقادیر FST و فاصله رینولدز62 استفاده شد.
از روش فاصله جفتی برای محاسبه مقادیر FST با 1000 جایگشت و مقدار=0 گاما اسفاده شد.
FS تست انتخاب بیطرفانه Fu برای تست گسترش جمعیتی اعمال شد.
حداقل ترین سطح معنی دار برای مقادیر FS بر میزان 0.02 تعیین شد (منبع: 63).
برای خلاصه نتایج آمار به جدول تکمیلی 7 مراجعه کنید.
آنالیز های ABC
برای کشف تاریخ دموگرافیک ساکاها ، با استفاده از 65،64BayeSSC چندین سناریوی کاندید فرمول بندی کردیم ، که پایه ای برای شبیه سازی نمونه های داده های ژنتیکی برای منطقه HVR-1 مهیا می کند.
محاسبات آمار خلاصه برای داده های مشاهده شده در DNaSP v5 صورت گرفت (منبع: 66).
برای تصدیق اینکه سناریو های کاندید توانایی تکثیر داده های ژنتیکی مشاهده شده را دارند ، ما توزیع پیشین از آمار خلاصه شبیه سازی شده را با مقادیر تجربی مشاهده شده قیاس کردیم.
همه آنالیز ها با پکیج abc در R 2.15.1 صورت گرفت (منابع: 67 ، 68).
ما به جزئیات کامل سناریو های دموگرافیک و آنالیز های اعمال شده در اینجا در یادداشت تکمیلی 1 می پردازیم.
آنالیز های ژنومی
آماده سازی کتابخانه DNA برای تعیین توالی متعاقب تفنگ ساچمه ای طبق پروتکل استفاده شده در Kircher 2012 (منبع: 69) با اصلاحات جزئی برای نمونه های تفنگ ساچمه ای صورت گرفت (یادداشت تکمیلی 3).
برای آنالیز داده های زنومی آمار های f3 و f4 محاسبه شد (qp3Pop و qpDstat از ADMIXTOOLS)37 ، آنالیز های جریان های تباری اعمال شد (qpWave/qpAdm)26 و آنالیز آمیختگی صورت گرفت39،38 (یادداشت تکمیلی 2).
دسترسی به داده ها
می توانید فایل های بم برای داده های ژنومی را از آرشیو نوکلئوتید اروپا بوسیله وارد کردن شماره PRJEB18686 بارگیری کنید.
توالی میتوکندری تحت شماره KY369766–KY369861 به ژن بانک سپرده شد.
منابع:
ref1 Parzinger, H. Die Skythen C.H. Beck (2004)
ref2 Herodotus, Herodotus: The History University of Chicago Press (1987)
ref3 Yablonsky, L. T. in Kurgans, Ritual Sites, and Settlements—Eurasian Bronze and Iron Age British Archaeological Reports (eds Davis-Kimball, J., Murphy, E. M., Koryakova, L. & Yablonsky, T.) 3-8 (Archaeopress, 2000)
ref4 Bashilov, V. A. & Yablonsky, L. T. in Kurgans, Ritual Sites, and Settlements—Eurasian Bronze and Iron Age British Archaeological Reports (eds Murphy Davis-Kimball, J., Koryakova, E. M. & Yablonsky, L. T.) 9-12 (Archaeopress, 2000)
ref5 Grjaznov, M. P. Der Großkurgan von Arzan in Tuva, Südsibirien Vol. 23, (C.H. Beck, 1984)
ref6 Alekseev, A. Y. et al. A chronology of the Scythian antiquities of Eurasia based on new archaeological and 14C data. Radiocarbon 43, 1085–1107 (2001)
ref7 Alekseev, A. Y. et al. Some problems in the study of the chronology of the ancient nomadic cultures in Eurasia (9th–3rd centuries BC). Geochronometria 21, 143–150 (2002)
ref8 Lalueza-Fox, C. et al. Unravelling migrations in the steppe: mitochondrial DNA sequences from ancient central Asians. Proc. R. Soc. Lond. B Biol. Sci. 271, 941–947 (2004)
ref9 Comas, D. et al. Trading genes along the silk road: mtDNA sequences and the origin of central Asian populations. Am. J. Hum. Genet. 63, 1824–1838 (1998)
CAS Article PubMed Central Google Scholar
ref10 Comas, D. et al. Admixture, migrations, and dispersals in Central Asia: evidence from maternal DNA lineages. Eur. J. Hum. Genet. 12, 495–504 (2004)
ref11 Quintana-Murci, L. et al. Where west meets east: the complex mtDNA landscape of the southwest and Central Asian corridor. Am. J. Hum. Genet. 74, 827–845 (2004)
CAS Article PubMed Central Google Scholar
ref12 Martinez-Cruz, B. et al. In the heartland of Eurasia: the multilocus genetic landscape of Central Asian populations. Eur. J. Hum. Genet. 19, 216–223 (2011)
ref13 Palstra, F. P., Heyer, E. & Austerlitz, F. Statistical inference on genetic data reveals the complex demographic history of human populations in Central Asia. Mol. Biol. Evol. 32, 1411–1424 (2015)
ref14 Chaix, R., Austerlitz, F., Hegay, T., Quintana-Murci, L. & Heyer, E. Genetic traces of east-to-west human expansion waves in Eurasia. Am. J. Phys. Anthropol. 136, 309–317 (2008)
ref15 Der Sarkissian, C. et al. Ancient DNA reveals prehistoric gene-flow from Siberia in the complex human population history of North East Europe. PLoS Genet. 9, e1003296 (2013)
CAS Article PubMed Central Google Scholar
ref16 Li, C. et al. Evidence that a west-east admixed population lived in the Tarim Basin as early as the early Bronze Age. BMC Biol. 8, 15 (2010)
Article PubMed Central Google Scholar
ref17 Allentoft, M. E. et al. Population genomics of Bronze Age Eurasia. Nature 522, 167–172 (2015)
ADS CAS Article Google Scholar
ref18 Ricaut, F. X., Keyser-Tracqui, C., Cammaert, L., Crubezy, E. & Ludes, B. Genetic analysis and ethnic affinities from two Scytho-Siberian skeletons. Am. J. Phys. Anthropol. 123, 351–360 (2004)
ref19 Pilipenko, A., Romaschenko, A., Molodin, V., Parzinger, H. & Kobzev, V. Mitochondrial DNA studies of the Pazyryk people (4th to 3rd centuries BC) from northwestern Mongolia. Archaeol. Anthropol. Sci. 2, 231–236 (2010)
ref20 Clisson, I. et al. Genetic analysis of human remains from a double inhumation in a frozen kurgan in Kazakhstan (Berel site, early 3rd century BC). Int. J. Legal. Med. 116, 304–308 (2002)
ref21 González-Ruiz, M. et al. Tracing the origin of the east-west population admixture in the Altai region (Central Asia). PloS ONE 7, e48904 (2012)
ADS Article PubMed Central Google Scholar
ref22 Samashev, Z. Berel’ (Almaty Publishing House ‘Taimas’, 2011)
ref23 Van Noten, F. & Polosmak, N. The frozen tombs of the Scythians. Endeavour 19, 76–82 (1995)
ref24 Rudenko, S. I. DrevnejŠie v mire chudoŽestvennye kovry i tkani, iz oledenelych kurganov gornogo Altaja (Iskusstvo, 1968)
ref25 Polosmak, N. V. et al. Textiles from the ‘Frozen’ Tombs in Gorny Altai 400–300 BC (An Integrated Study) Siberian Branch of the Russian Academy of Sciences (2006)
ref26 Haak, W. et al. Massive migration from the steppe was a source for Indo-European languages in Europe. Nature 522, 207–211 (2015)
ADS CAS Article PubMed Central Google Scholar
ref27 Fu, Q. et al. An early modern human from Romania with a recent Neanderthal ancestor. Nature 524, 216–219 (2015)
ADS CAS Article PubMed Central Google Scholar
ref28 Beaumont, M. A., Zhang, W. & Balding, D. J. Approximate Bayesian computation in population genetics. Genetics 162, 2025–2035 (2002)
PubMed PubMed Central Google Scholar
ref29 Kohl, P. L. The Making of Bronze Age Eurasia Cambridge University Press (2007)
ref30 Molodin, V. I. et al. in Population Dynamics in Prehistory and Early History—New Approaches Using Stable Isotopes and Genetics (eds Burger, J., Kaiser, E.& Schier, W.) 93-112 (De Gruyter, 2012)
ref31 Keyser, C. et al. Ancient DNA provides new insights into the history of south Siberian kurgan people. Hum. Genet. 126, 395–410 (2009)
ref32 Molodin, V. I., Pilipenko, A. S., Zhuravlev, A. A., Trapezov, R. O. & Romashchenko, A. G. An analysis of mitochondrial DNA from the Pakhomovskaya population of the late Bronze Age, Western Siberia. Archaeol. Ethnol. Anthropol. Eurasia 40, 62–69 (2012)
ref33 Patterson, N., Price, A. L. & Reich, D. Population structure and eigenanalysis. PLoS Genet. 2, e190 (2006)
Article PubMed Central Google Scholar
ref34 Mathieson, I. et al. Genome-wide patterns of selection in 230 ancient Eurasians. Nature 528, 499–503 (2015)
ADS CAS Article PubMed Central Google Scholar
ref35 Lazaridis, I. et al. Ancient human genomes suggest three ancestral populations for present-day Europeans. Nature 513, 409–413 (2014)
ADS CAS Article PubMed Central Google Scholar
ref36 Jones, E. R. et al. Upper palaeolithic genomes reveal deep roots of modern Eurasians. Nat. Commun. 6, 8912 (2015)
CAS Article PubMed Central Google Scholar
ref37 Patterson, N. et al. Ancient admixture in human history. Genetics 192, 1065–1093 (2012)
Article PubMed Central Google Scholar
ref38 Alexander, D. H., Novembre, J. & Lange, K. Fast model-based estimation of ancestry in unrelated individuals. Genome Res. 19, 1655–1664 (2009)
CAS Article PubMed Central Google Scholar
ref39 Alexander, D. & Lange, K. Enhancements to the ADMIXTURE algorithm for individual ancestry estimation. BMC Bioinformatics 12, 246 (2011)
Article PubMed Central Google Scholar
ref40 Fu, Q. et al. Genome sequence of a 45,000-year-old modern human from western Siberia. Nature 514, 445–449 (2014)
ADS CAS Article PubMed Central Google Scholar
ref41 Seguin-Orlando, A. et al. Genomic structure in Europeans dating back at least 36,200 years. Science 346, 1113–1118 (2014)
ADS CAS Article Google Scholar
ref42 Raghavan, M. et al. Upper palaeolithic Siberian genome reveals dual ancestry of native Americans. Nature 505, 87–91 (2014)
ref43 Soejima, M., Tachida, H., Ishida, T., Sano, A. & Koda, Y. Evidence for recent positive selection at the human AIM1 locus in a European population. Mol. Biol. Evol. 23, 179–188 (2006)
ref44 Sabeti, P. C. et al. Genome-wide detection and characterization of positive selection in human populations. Nature 449, 913–918 (2007)
ADS CAS Article PubMed Central Google Scholar
ref45 Wilde, S. et al. Direct evidence for positive selection of skin, hair, and eye pigmentation in Europeans during the last 5,000 y. Proc. Natl Acad. Sci. USA 111, 4832–4837 (2014)
ADS CAS Article Google Scholar
ref46 Han, Y. et al. Evidence of positive selection on a class I ADH locus. Am. J. Hum. Genet. 80, 441–456 (2007)
CAS Article PubMed Central Google Scholar
ref47 Peng, Y. et al. The ADH1B Arg47His polymorphism in East Asian populations and expansion of rice domestication in history. BMC Evol. Biol. 10, 15 (2010)
Article PubMed Central Google Scholar
ref48 McVean, G. A. et al. An integrated map of genetic variation from 1,092 human genomes. Nature 491, 56–65 (2012)
ADS CAS Article Google Scholar
ref49 Hollard, C. et al. Strong genetic admixture in the Altai at the Middle Bronze Age revealed by uniparental and ancestry informative markers. Forensic Sci. Int. Genet. 12, 199–207 (2014)
ref50 Hellenthal, G. et al. A genetic atlas of human admixture history. Science 343, 747–751 (2014)
ADS CAS Article PubMed Central Google Scholar
ref51 Torroni, A. et al. Native American mitochondrial DNA analysis indicates that the Amerind and the Nadene populations were founded by two independent migrations. Genetics 130, 153–162 (1992)
CAS PubMed PubMed Central Google Scholar
ref52 Finnilä, S., Lehtonen, M. S. & Majamaa, K. Phylogenetic network for European mtDNA. Am. J. Hum. Genet. 68, 1475–1484 (2001)
Article PubMed Central Google Scholar
ref53 Kivisild, T. et al. The emerging limbs and twigs of the East Asian mtDNA tree. Mol. Biol. Evol. 19, 1737–1751 (2002)
ref54 Meyer, M., Stenzel, U. & Hofreiter, M. Parallel tagged sequencing on the 454 platform. Nat. Protoc. 3, 267–278 (2008)
ref55 Niu, B., Fu, L., Sun, S. & Li, W. Artificial and natural duplicates in pyrosequencing reads of metagenomic data. BMC Bioinformatics 11, 187 (2010)
Article PubMed Central Google Scholar
ref56 Katoh, K., Misawa, K., Kuma, K. & Miyata, T. MAFFT: a novel method for rapid multiple sequence alignment based on fast Fourier transform. Nucleic Acids Res. 30, 3059–3066 (2002)
CAS Article PubMed Central Google Scholar
ref57 Kloss-Brandstätter, A. et al. HaploGrep: a fast and reliable algorithm for automatic classification of mitochondrial DNA haplogroups. Hum. Mutat. 32, 25–32 (2011)
ref58 van Oven, M. & Kayser, M. Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation. Hum. Mutat. 30, E386–E394 (2009)
ref59 Excoffier, L. & Lischer, H. E. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows. Mol. Ecol. Resour. 10, 564–567 (2010)
ref60 Nei, M. Molecular Evolutionary Genetics Columbia University Press (1987)
ref61 Tajima, F. Evolutionary relationship of DNA sequences in finite populations. Genetics 105, 437–460 (1983)
CAS PubMed PubMed Central Google Scholar
ref62 Reynolds, J., Weir, B. S. & Cockerhem, C. C. Estimation of the coancestry coefficient: basis for a short-term genetic distance. Genetics 105, 767–779 (1983)
CAS PubMed PubMed Central Google Scholar
ref63 Fu, Y. X. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection. Genetics 147, 915–925 (1997)
CAS PubMed PubMed Central Google Scholar
ref64 Anderson, C. N., Ramakrishnan, U., Chan, Y. L. & Hadly, E. A. Serial SimCoal: a population genetics model for data from multiple populations and points in time. Bioinformatics 21, 1733–1734 (2005)
ref65 Excoffier, L., Novembre, J. & Schneider, S. SIMCOAL: a general coalescent program for the simulation of molecular data in interconnected populations with arbitrary demography. J. Hered. 91, 506–509 (2000)
ref66 Librado, P. & Rozas, J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data. Bioinformatics 25, 1451–1452 (2009)
ref67 Csillery, K., Francois, O. & Blum, M. G. B. abc: an R package for approximate Bayesian computation (ABC). Methods Ecol. Evol. 3, 475–479 (2012)
ref68 R_Core_Team. R: A language and environment for statistical computing. http://www.R-project.org/ (2013)
ref69 Kircher, M., Sawyer, S. & Meyer, M. Double indexing overcomes inaccuracies in multiplex sequencing on the Illumina platform. Nucleic Acids Res. 40, e3 (2012)
سپاسگزاری ها
از لئونید یابلونسکی بخاطر ارائه نمونه های پکروفکا تشکر می کنیم و همچنین از کمک های خانم جنین دویس کیمبال در طی فرایند نمونه برداری سپاسگزاریم.
از خانم ماریا مدنیکووا و خانم ماریا دوبروولسکایا برای تهیه نمونه ها به ترتیب از نووزاودنو و کلبینو سپاسگزاریم.
از آنتون گاس و آناتولی نگلر بخاطر اطلاعات اضافی باستان شناسی سپاسگزاریم.
بعضی از بررسی های محاسباتی فشرده بر خوشه لینوکس ‘موزه ملی تاریخ طبیعی’ در پاریس (تحت مدیریت جولیو پدرازا آکوستا) انجام شده است.
دیوید رایش توسط کمک NIH GM100233 ، و توسط NSF HOMINID BCS-1032255 حمایت شد ، ایشان یک پژوهشگر در انستیتو پزشکی هاورد هیوز هستند.
این کار توسط وزارت آموزش و پژوهش فدرال آلمان حمایت شد.
ماتیاس کورات توسط کمک 31003A_156853 بنیاد ملی علوم سوئیس حمایت شد.
آزمایش های DNA باستان در آزمایشگاه نووسیبیرسک توسط بنیاد علوم روسیه (RSCF) ، (پروژه شماره 14-50-00036) حمایت مالی شد.
اطلاعات نویسنده
مارتینا اونترلاندر و فریزو پالسترا: این دو نویسنده به طور مساوی در این کار نقش داشته اند.
وابستگی ها
گروه پالئوزنتیک ، انستیتو زیست شناسی تکاملی ، دانشگاه یوهانس گوتنبرگ ماینتس ، ماینتس ، 55099 ، آلمان
مارتینا اونترلاندر ، زوزانا هافمنووا ، ملانی گروس ، کریستیان سل ، جنس بلوچر ، کارولا کیرسانوو ، ساندرا وایلد ، آدام پاول و یواخیم برگر
مرکز ملی پژوهش های علمی واحد تحقیقات مشترک 7206 انسان شناسی-محیط زیست ، موزه ملی تاریخ طبیعی ، پاریس ، 75016 ، فرانسه
فریزو پالسترا ، میریام ژرژ و اویلین هایر
بخش ژنتیک ، مدرسه پزشکی هاروارد ، بوستون ، 02115 ، ماساچوست ، ایالات متحده آمریکا
یوسف لازاریدیس ، نادین رولند و دیوید رایش
انستیتو Broad ام آی تی و هاروارد ، کمبریج ، 02142 ، ماساچوست ، ایالات متحده آمریکا
یوسف لازاریدیس و دیوید رایش
انستیتو سیتولوژی و ژنتیک ، شعبه سیبری ، آکادمی علوم روسیه ، آکادمیک لاورنتیف 10 ، نووسیبیرسک ، 630090 ، روسیه
الکساندر پیلیپنکو ، دیمیتری پوزدنیاکف و ویاچسلاو آی. مولودین
انستیتو باستان شناسی و قوم نگاری ، شعبه سیبری ، آکادمی علوم روسیه ، آکادمیک لاورنتیف 17 ، نووسیبیرسک ، 630090 ، روسیه
الکساندر پیلیپنکو و ویاچسلاو آی. مولودین
دانشگاه ایالتی نووسیبیرسک ، خیابان پیروگووا 2 ، نووسیبیرسک ، 630090 ، روسیه
الکساندر پیلیپنکو
گروه ژنتیک مولکولی و آنالیز ژنوم ، انستیتو زیست شناسی تکاملی ، دانشگاه یوهانس گوتنبرگ ماینتس ، ماینتس ، 55099 ، آلمان
بنجامین ریگر
بخش تاریخ و مطالعات فرهنگی ، دانشگاه آزاد برلین ، برلین ، 14195 ، آلمان
الکه کایسر و ولفرام شییر
دانشگاه ایالتی علوم اجتماعی و آموزش سامارا ، سامارا ، 443099 ، فدراسیون روسیه
الکساندر خخلف
انستیتو مکس پلانک برای علوم تاریخ انسان ، خیابان کاتولیک 10 ، ینا ، 07745 ، آلمان
آدام پاول
بخش ژنتیک و تکامل ، واحد انسان شناسی ، دانشگاه ژنو ، ژنو ، 1205 ، سوئیس
ماتیاس کورات
انستیتو پزشکی هاورد ، مدرسه پزشکی هاروارد ، بوستون ، 02115 ، ماساچوست ، ایالات متحده آمریکا
دیوید رایش
انستیتو باستان شناسی شعبه مارگولان ، آستانا ، 010000 ، قزاقستان
زینولا ساماشف
بنیاد میراث فرهنگی پروس ، برلین ، 10785 ، آلمان
هرمان پارزینگر
مشارکت ها
یواخیم برگر و هرمان پارزینگر ، طراحی پژوهش.
الکه کایسر ، ولفرام شییر ، دیمیتری پوزدنیاکف ، الکساندر خخلف ، هرمان پارزینگر ، زینولا ساماشف و ویاچسلاو آی. مولودین ، ارائه متریال نمونه ، اطلاعات پس زمینه باستان شناسی و/یا اطلاعات انسان شناسی.
مارتینا اونترلاندر و الکساندر پیلیپنکو ، پردازش نمونه های DNA باستانی و ارائه توالی میتوکندری.
ملانی گروس و جنس بلوچر ، پردازش نمونه های DNA باستانی و آماده سازی کتابخانه های NGS.
زوزانا هافمنووا ، ملانی گروس ، کریستیان سل ، جنس بلوچر و ماتیاس کورات ، آنالیز داده های تفنگ ساچمه ای.
یوسف لازاریدیس ، نادین رولند و دیوید رایش ، انجام ظبط هسته و متعاقبا آنالیز ژنومی بر داده های SNP.
فریزو پالسترا ، طراحی و انجام آنالیز های ABC بر mtDNA.
مارتینا اونترلاندر ، زوزانا هافمنووا ، فریزو پالسترا ، آدام پاول ، ماتیاس کورات ، هرمان پارزینگر و یواخیم برگر ، تفسیر نتایج و قرار دهی در زمینه.
مارتینا اونترلاندر ، کارولا کیرسانوو و ساندرا وایلد ، پردازش آنالیز های SNP فنوتیپی بر 454 داده.
بنجامین ریگر ، ارائه کد برای آنالیز های 454 داده.
میریام ژرژ و اویلین هایر ، آنالیز و ارائه داده mtDNA معاصر از آسیای میانه.
مارتینا اونترلاندر ، فریزو پالسترا ، یوسف لازاریدیس ، کارولا کیرسانوو ، دیوید رایش و یواخیم برگر ، تالیف مقاله با کمک نویسندگان مشترک.
نویسنده مسئول مکاتبه
مکاتبه با یواخیم برگر.
PDF اطلاعات تکمیلی شامل: شکل های تکمیلی 1-14 ، جداول تکمیلی 1-28 ، یادداشت تکمیلی 1 و منابع تکمیلی (حجم فایل: PDF 5127 کیلوبایت).
بارگیری PDF پرونده مرور (حجم فایل: 1519 کیلوبایت).
جزئیات از PDF اطلاعات تکمیلی
قسمتی از PDF اطلاعات تکمیلی برای شرح جزئیات ارتباط ساکاها با جمعیت های معاصر (شامل شکل های تکمیلی 10 ، 11 و جدول تکمیلی 19).
| A نیا از ساکاهای غربی (قرمز تیره)
شکل تکمیلی 10. احتمالات پسین چهار سناریوی کاندید برای ارتباط 86 جمعیت معاصر اوراسیا با ساکاهای عصر آهن. هر پنل مدل های پسین را به ترتیب (A نیا از ساکاهای غربی (قرمز تیره) ، (B نیا از ساکاهای شرقی (خاکستری تیره) ، (C رابطه نیایی با ساکاهای غربی و (D رابطه نیایی با ساکاهای شرقی (سبز تیره) نشان می دهد. رنگ سناریو های دیگر برای کمک به شرح کم رنگ تر هستند. خطوط افقی در میان تصویر مقادیر 50% و 90% را برای مدل احتمال پسین نشان می دهند. برای شرح کد جمعیت ها در سمت چپ تصویر ، جدول تکمیلی 19 را ببینید.
|
| B نیا از ساکاهای شرقی (خاکستری تیره)
ادامه شکل تکمیلی 10.
|
| C رابطه نیایی با ساکاهای غربی (سیاه)
ادامه شکل تکمیلی 10.
|
| D رابطه نیایی با ساکاهای شرقی (سبز تیره)
ادامه شکل تکمیلی 10.
|
|
شکل تکمیلی 11. مدل پسین برای نیا از جمعیت های ساکایی برای 86 جمعیت معاصر در آسیای میانه. برای هر یک از نمونه های معاصر نمودار دایره ای رسم شده است که نشان دهنده مدل های پسین برای نیا از ساکاهای غربی (سیاه) ، نیا از ساکاهای شرقی (خاکستری) ، رابطه نیایی با ساکاهای غربی (قرمز) ، رابطه نیایی با ساکاهای شرقی (سبز) است. همچنین (منطقه نارنجی) نیز مکان تقریبی نمونه DNA های باستانی و محدوده تاریخی ایل های ساکایی را نشان می دهد. برای اطلاعات مشروح درباره جمعیت های معاصر به جدول تکمیلی 19 مراجعه کنید. منبع: نقشه بنیادی توسط تام پترسون ، بارگیری شده از www.shadedrelief.com.
|
جدول تکمیلی 19. مشخصات نمونه 86 جمعیت معاصر استپ اوراسیا. برای هر جمعیت اطلاعاتی چون مکان نمونه ، کد ، اندازه نمونه (S) و منبع نشر اصلی ارائه شده است. همچنین آمار های خلاصه درون جمعیتی مانند تعداد هاپلوتیپ (Nh) ، میاگین تعداد تفاوت های جفتی (k) ، تنوع نوکلئوتیدی (π) و تاجیما (D) نیز در جدول گنجانده شده است.
برگردان فارسی و تدوین: حسین خیری ، نشر الکترونیک: ائل اوبا
استناد:
Unterländer, M. et al. Ancestry and demography and descendants of Iron Age nomads of the Eurasian Steppe. Nat. Commun. 8, 14615 doi: 10.1038/ncomms14615 (2017)



مطالب مرتبط
چگونه تهران پایتخت شد؟
معرفی سکاها
اولین های کتب درسی در دارالفنون